No proceso de realizar estudos xenéticos, adoitamos atopar mostras de ARN insuficientes, por exemplo, para estudar pequenos tumores anatómicos orais, incluso mostras unicelulares e mostras de mutacións xenéticas específicas que se transcriben en niveis moi baixos nas células humanas.Iso si, para a proba do COVID-19, se os hisopos non se atopan no lugar axeitado ou non son suficientes veces durante a toma de mostras, o tamaño da mostra será moi baixo, polo que a Comisión de Sanidade e Ordenación Familiar saíu hai dous días e pasou a proba, e se o mostrador de ácido nucleico non tomou seis mostras, pode informar.
A sensibilidade do reactivo é importante porque temos este ou ese problema, entón que podemos facer para mellorar a sensibilidade da RT-PCR?
Antes de discutir posibles solucións, mencionemos dúas grandes complicacións coa situación que acabamos de mencionar.
En primeiro lugar, preocúpanos a perda de ARN cando só temos unhas poucas poboacións celulares na nosa mostra.Se se utilizan métodos tradicionais de separación e limpeza, como o método da columna ou o método de precipitación de ácidos nucleicos, existe unha gran posibilidade de que se perdan as poucas mostras.Unha solución é engadir unha molécula portadora, como o ARNt, pero aínda así, non hai garantía de que o noso experimento de recuperación estea correcto.
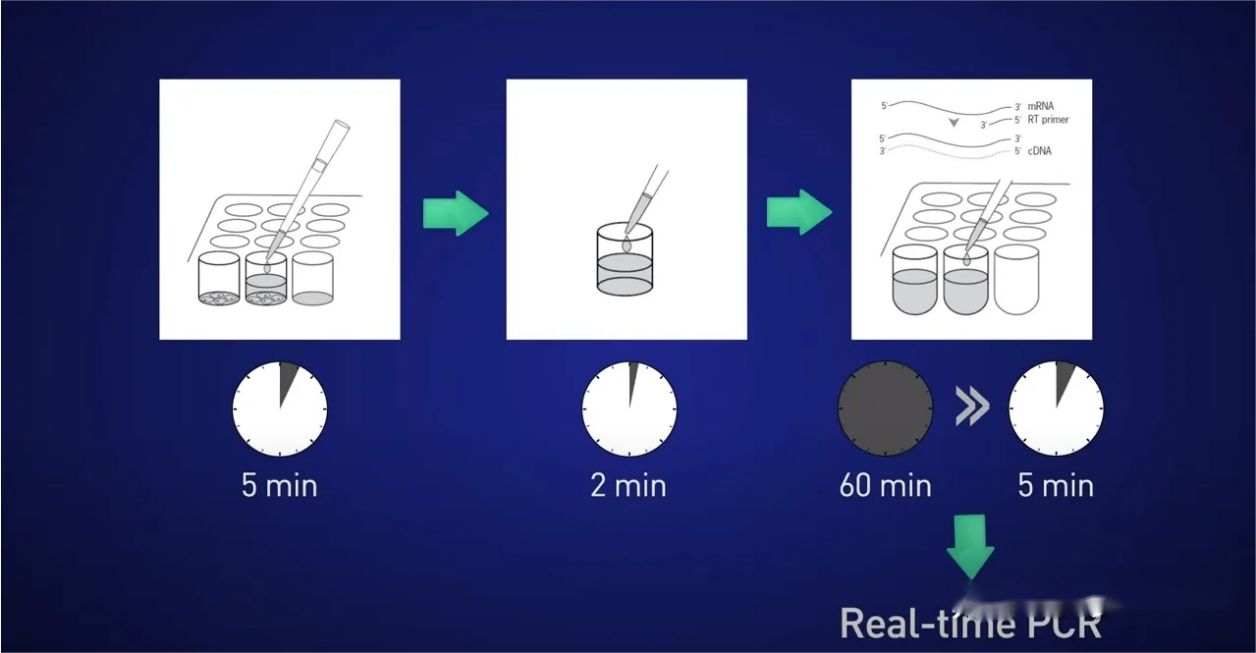
Entón, cal é mellor?Unha boa opción para células cultivadas ou mostras microanatómicas é utilizar a lise directa.
A idea é dividir as células durante 5 minutos, liberar o ARN na solución, despois deter a reacción durante 2 minutos, engadir o lisado directamente á reacción de transcrición inversa para que non se perda ARN e, finalmente, poñer directamente o ADNc resultante. na reacción en tempo real.
Pero e se, por mor dun punto de partida limitado ou dunha pequena cantidade de expresión xenética diana, podemos reciclar todo o ARN e aínda non proporcionar modelos suficientes para obter un bo sinal en tempo real?
Neste caso, o paso de preamplificación pode ser moi útil.
O seguinte é un esquema para aumentar a sensibilidade despois da transcrición inversa.Antes de comezar, cómpre preguntar augas abaixo que dianas nos interesan, para deseñar cebadores específicos para estas dianas para a súa preamplificación.
Isto pódese conseguir creando un cebador mixto con ata 100 pares de cebadores e un ciclo de reacción de 10 a 14 veces.Polo tanto, é necesario un Master Mix deseñado especificamente para este requisito para preamplificar o ADNc obtido.
O motivo para establecer o número de ciclos entre 10 e 14 é que este número limitado de ciclos garante a aleatoriedade entre os distintos obxectivos, o que é crucial para os investigadores que necesitan información molecular cuantitativa.
Despois da preamplificación, podemos obter unha gran cantidade de ADNc, de xeito que a sensibilidade de detección no back-end mellore moito, e incluso podemos diluír a mostra e realizar varias reaccións de PCR en tempo real para eliminar posibles erros aleatorios.
Hora de publicación: 11-Abr-2023